Секвенирование нуклеиновых кислот
- Главная страница
- Пресс-центр
- Статьи
- Секвенирование нуклеиновых кислот
Секвенирование нуклеиновых кислот — это процесс определения последовательности нуклеотидов в цепочке ДНК.
Основными задачами генетического анализа являются:
- обнаружение неизвестных ранее последовательностей нуклеиновых кислот, например, геномов нового типа;
- распознавание уникальных особенностей конкретного образца среди обобщённой последовательности (ресеквенирование);
- исследование генетического разнообразия, в том числе однонуклеотидных замен (SNP-типирование);
- определение эпигенетических модификаций, например, профиля метилирования;
- изучение профиля экспрессии отдельно взятых клеток секвенированием кДНК, полученной из тотальной мРНК клетки.
Сравнительные таблицы производительности секвенаторов
Капиллярные
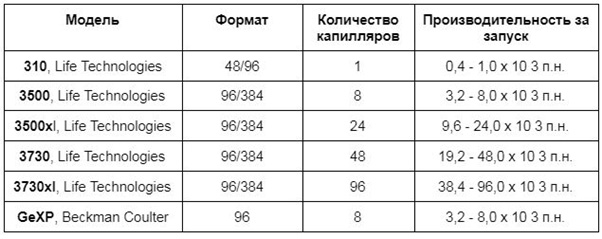
Пиросеквенаторы

NGS-секвенаторы:
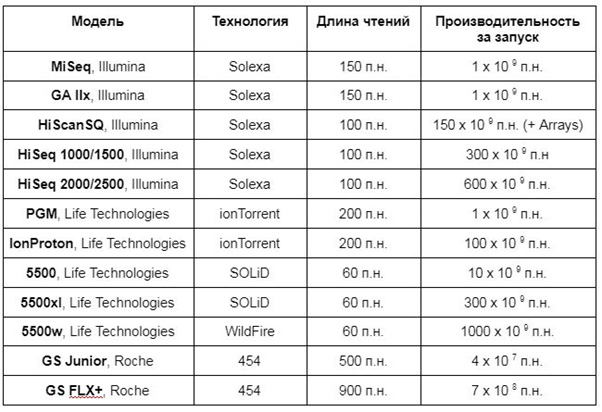
Генетические анализаторы различаются по ряду характеристик, среди которых:
- Длина прочтений.
- короткие;
- средние;
- длинные.
- Применяемая технология.
- технология по методу Сэнгера;
- пиросеквенирование;
- ионное полупроводниковое секвенирование;
- циклическое лигазное секвенирования;
- секвенирования на молекулярных кластерах с использованием флуоресцентно меченых нуклеотидов;
- одномолекулярное секвенирование.
- Число прочтений за запуск.
- Количество анализируемых одновременно маркеров.
- Время, необходимое для запуска.
- Стоимость самого прибора, одного запуска, анализа одной пробы.
Технологии секвенирования
На сегодняшний день существуют несколько основных методов считывания последовательности ДНК при помощи секвенаторов — классические и нового поколения.
Метод Сэнгера (ферментативный)
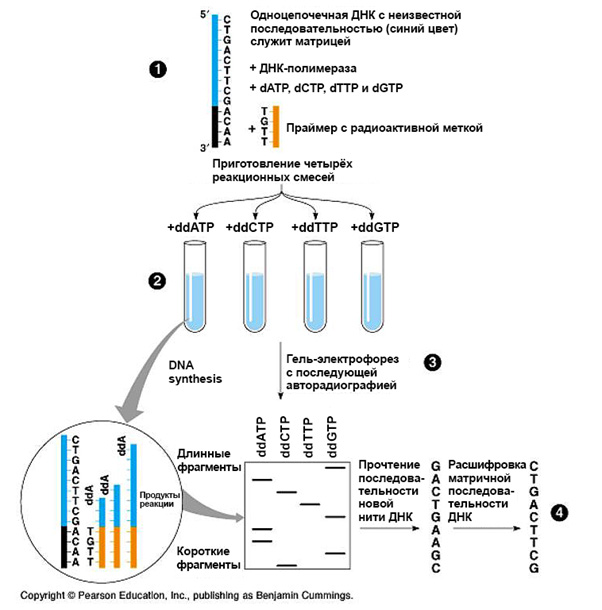
Секвенирование по Сэнгеру, также известное как метод «обрыва цепи», было разработано и предложено биохимиком Фредериком Сэнгером в 1977 году. Суть заключается в синтезе новой комплементарной ДНК-цепи на ДНК-матрице с помощью фермента ДНК-полимеразы.
Синтез проводится в 4 разных пробирках с реакционной смесью, состоящей из праймера, стандартных дезоксинуклеотидов (dATP, dGTP, dCTP, dTTP), а также малого количества одного из четырёх радиоактивно меченых дезоксинуклеотидов (дидезоксинуклеотидов). Готовятся четыре раствора с каждым ddNTP по отдельности. У дидезоксирибонуклеотидов (ddATP, ddGTP, ddCTP, or ddTTP) отсутствует 3'-гидроксильная группа, поэтому после их включения в цепь дальнейший синтез обрывается.
Таким образом, в каждой пробирке образуется набор фрагментов ДНК разной длины, которые заканчиваются одним и тем же нуклеотидом (в соответствии с добавленным дидезоксинуклеотидом). После завершения реакции содержимое пробирок разделяют электрофорезом в полиакриламидном геле в денатурирующих условиях и проводят авторадиографию гелей.
На сегодняшний день секвенирование ДНК по Сэнгеру полностью автоматизировано. В настоящее время вместо радиоактивно меченных нуклеотидов используют дидезоксинуклеотиды с флуоресцентными метками с разными длинами волн испускания, благодаря этому реакцию можно проводить в одной пробирке.
Реакционную смесь разделяют капиллярным электрофорезом, фрагменты ДНК, выходящие из капиллярной колонки, регистрируются детектором флуоресценции. Результаты анализируют с помощью компьютера и представляют в виде последовательности разноцветных пиков, соответствующих четырём нуклеотидам. Секвенаторы такого типа могут «прочитывать» за один раз последовательности длиной 500-1000 нуклеотидов.
Циклическое лигазное секвенирование
Технология циклического лигазного секвенирования (Sequencing by Oligonucleotide Ligation and Detection — SOLiD) разработана компанией Life Technologies и является коммерчески доступной с 2006 года. Этот метод можно разделить на следующие этапы: подготовка библиотек, ПЦР в эмульсии, насыщение бусин целевой ДНК, перенос бусин, секвенирование, расшифровка данных.
На первом этапе целевая ДНК разрезается на небольшие фрагменты, а затем к фрагментам пришиваются короткие нуклеотидные последовательности адаптеры А1 и А2. В итоге в библиотеке оказываются одноцепочечные нуклеотидные последовательности A1-(фрагмент ДНК)-A2.
После этого проводят ПЦР ДНК-фрагментов библиотеки в небольших каплях. Каждая капля содержит праймеры P1 и Р2, которые являются комплементарными по отношению к фрагментам А1 и А2. Каждая бусина несёт только один фрагмент праймера. В результате чего на каждой бусине оказывается по одной цепочке ДНК комплементарной исходному образцу.
Секвенирование проходит с помощью лигирования восьми-нуклеотидных зондов, меченных на 5’-конце одним из четырех различных флуорофоров. Последовательность зондов несёт сайт гидролиза, находящийся между пятым и шестым нуклеотидами. Первые два основания (считая с 3’-конца) комплементарны двум нуклеотидам секвенируемой последовательности.
С третьего по пятый основания зонда могут гибридизоваться с любыми тремя нуклеотидами секвенируемой ДНК. 6-8 основания зонда также могут гибридизоваться с любой последовательностью, однако они вместе с флуоресцентным красителем отщепляются от зонда в ходе реакции.
Отщепление флуоресцентной метки вместе с основаниями 6-8 происходит таким образом, что на 5'-конце зонда остается фосфатная группа, способствующая следующему циклу лигирования зонда. Так, два основания каждого зонда точно комплементарны основаниям секвенируемой последовательности в позициях n+1 и n+2, n+6 и n+7 и т.д.
На выходе мы получаем результаты по флуоресценции. Пространство цветов и пространство нуклеотидов содержат по 4 элемента. Каждый цвет кодирует собой 4 из 16 возможных динуклеотидов. Например, «синий» цвет флуоресцентной метки соответствует паре одинаковых нуклеотидов (то есть AA,GG,TT или CC). Дизайн матрицы преобразований цвета способствует коррекции ошибок.
Главным преимуществом этого метода является то, что каждый нуклеодит прочитывается дважды, что сильно повышает точность секвенирования.
Метод пиросеквенирования
Технология была разработана Полом Ниреном и его студентом Мустафой Ронаги в 1996 году. Она основывается на синтезе новой ДНК-цепи на иммобилизированной анализируемой матрице с участием полимеразы. В качестве затравки выступает праймер. Затем происходит поочерёдное добавление каждого типа дезоксинуклеотидтрифосфатов (A, G, C, T).
При встраивании нуклеотида стехиометрически высвобождается побочный продукт — пирофосфат (PPi). Он, в свою очередь, с участием фермента ATP-сульфурилазы в присутствии аденозин-5'-фосфосульфата (dATPαS) превращается в ATP. ATP — источник энергии для фермента люциферазы, превращающей люциферин в оксилюциферин с выделением света. Компьютерная программа осуществляет детекцию и анализ света.
Невовлечённые в синтез нуклеотиды и ATP под воздействием фермента апиразы распадаются, после чего запускается следующий цикл реакции, с новым нуклеотидом.
Ионное полупроводниковое секвенирование
Данная технология является методом определения последовательности ДНК, основанным на обнаружении ионов водорода, которые выделяются во время полимеризации ДНК. Это метод «секвенирования при синтезе», в ходе которого комплементарная цепь строится на основе последовательности матричной цепи.
Микролунки, содержащие предназначенную для секвенирования молекулу матричной ДНК, наполняют дезоксирибонуклеотидтрифосфатом (dNTP) одного вида. Если введенный dNTP является комплементарным к ведущему нуклеотиду матрицы, он включается в растущую комплементарную цепь.
Это вызывает высвобождение ионов водорода, который вызывает срабатывание ионного датчика, который указывает, что реакция произошла. Если в последовательности матричной цепи присутствует повтор одного нуклеотида, несколько молекул dNTP будут присоединены в одном цикле. Это приводит к увеличению количества образовавшихся ионов водорода и пропорционально более высокому электрическому сигналу.
Эта технология отличается от других технологий секвенирования тем, что не использует модифицированные нуклеотиды и оптические датчики. Ion semiconductor sequencing может также упоминаться как Ion torrent sequencing, рН-опосредованное секвенирование, или полупроводниковое секвенирование.
Технология ионного полупроводникового секвенирования позволяет исследовать большие объёмы данных, например: транскриптомы отдельных клеток и тканей, метагеномы микробных сообществ, малые РНК и проводить ChIP-seq.
Одномолекулярное секвенирование
Данный метод разработан компанией Pacific BioScinces. Идея метода состоит в определении последовательности ДНК за счёт наблюдения за работой единичной молекулы ДНК-полимеразы в реальном времени.
При этом ДНК-полимераза достраивает вторую цепь исследуемой молекулы ДНК, используя нуклеотиды, меченные различными флуорофорами; регистрируя данные метки, можно понять, какой нуклеотид ДНК-полимераза встраивает в настоящий момент.
Этот метод секвенирования позволяет получать очень длинные прочтения порядка десятков тысяч нуклеотидов. Кроме того, метод работает без предварительной амплификации образца посредством ПЦР. Одномолекулярное секвенирование работает с минимальным количество исходной ДНК.
Бисульфитный метод
Это название группы методов, в основе которых один принцип: определение паттерна метилирования ДНК посредством её бисульфитной обработки.
Под воздействием бисульфита остатки цитозина превращаются в остатки урацила, но при этом не затрагиваются метилированные остатки 5-метилцитозина. Далее изменённая последовательность сравнивается с исходной, цитозины и тимины разделяются в изученных последовательностях, и можно установить, какие CpG-динуклеотиды были метилированы.
и приглашать на предстоящие выставки и семинары


